การแปลง KMD ฐานเศษส่วนสำหรับโพลีเอสเตอร์โพลี (3-ไฮดรอกซีบิวทีเรต) ที่มีน้ำหนักโมเลกุลสูงหลังจากการย่อยสลายด้วยอัลคาไลน์บนเพลตและการวิเคราะห์ SpiralTOF™" [แอปพลิเคชัน MALDI]
MSTips หมายเลข 283
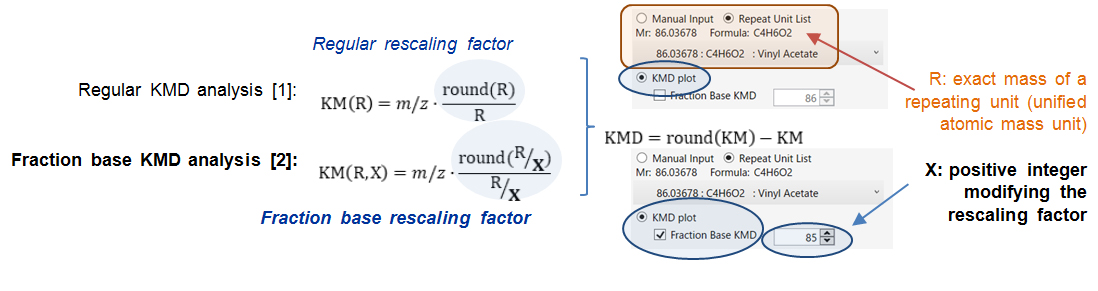
บทนำ: ภูมิหลังทางคณิตศาสตร์
โพลิเมอร์ที่มีน้ำหนักโมเลกุลสูงมักเป็น MS-silent เนื่องจากมีการกระจายตัวสูงโดยธรรมชาติ (ĐM) หรือตรวจจับได้ในช่วงมวลสูงที่มีกำลังแยกต่ำ ดังนั้น การย่อยสลายด้วยด่างแบบ "บนจาน" จึงได้รับการพัฒนาเป็นตัวอย่างการปรับสภาพล่วงหน้าบนเป้าหมาย MALDI ด้วยโพลิเมอร์ 1 ใน 3 ของ ng เพื่อตัดโซ่โพลีเอสเตอร์อุตสาหกรรมขนาดยาวให้เป็นโอลิโกเมอร์สั้นที่คล้อยตาม MALDI-HRMS [2] สเปกตรัมมวลที่ซับซ้อนของโอลิโกเมอร์ P3HB ได้รับการวิเคราะห์โดยพล็อตฐานเศษส่วน Kendrick Mass Defect (KMD) [XNUMX] การวิเคราะห์ KMD ฐานเศษส่วนได้รับการพัฒนาจากการวิเคราะห์ KMD ปกติเพื่อปรับเปลี่ยนลักษณะและความสามารถในการแยกของแปลง KMD ขึ้นอยู่กับตัวหาร (หมายเหตุ X) นอกเหนือจากหน่วยฐาน (หมายเหตุ R) ผ่านฐานเศษส่วน R/X [XNUMX ].
การทดลอง
โพลี(3-ไฮดรอกซีบิวทิเรต) (P3HB, Mn = 2.6 × 105 กรัม โมล-1, ĐM=2.7) มาจาก Sigma-Aldrich (เซนต์หลุยส์ มิสซูรี สหรัฐอเมริกา) สารเคมีและตัวทำละลายอื่นๆ ทั้งหมดมาจาก FUJIFILM Wako Pure Chemical Corporation (โอซาก้า ประเทศญี่ปุ่น) เว้นแต่จะระบุไว้เป็นอย่างอื่น 1 ไมโครลิตรของ P3HB ใน THF ที่ 1 มก. มล-1 ถูกโยนทิ้งบนแผ่นเป้า MALDI แบบใช้แล้วทิ้ง (Hudson Surface Technology, New Jersey, USA) เพื่อสร้างชั้นโพลิเมอร์บางๆ โซเดียมไฮดรอกไซด์ 1 ไมโครลิตรในเมทานอล (10 มก. มล-1) ถูกวางบนชั้นตัวอย่างและทิ้งไว้ให้แห้งเป็นเวลา 5 นาที น้ำยาอัลคาไลน์ส่วนเกินถูกล้างด้วยน้ำกลั่น หลังจากการอบแห้ง 1 ไมโครลิตรของ 2,4,6-trihydroxyacetophenone (THAP, Protea Biosciences, West Virginia, USA) ใน THF (20 มก. มล.-1) ถูกนำไปใช้กับจุดตัวอย่างเป็นเมทริกซ์ แมสสเปกตรัมถูกบันทึกด้วยแมสสเปกโตรมิเตอร์ JMS-S3000 SpiralTOF™ แปลงคำนวณโดยใช้ msRepeatFinder 3.0 จากรายการสูงสุดที่ส่งออกโดยใช้ msTornado™
การวิเคราะห์มวลของ MALDI
ชุดไอออนเข้มข้นของ P3HB oligomers ถูกพบใน MALDI-MS (รูปที่ 1A) ตรวจพบชุดข้อมูลหลัก 1 ชุดที่แตกต่างกันไปตามกลุ่มปลายทาง (รูปที่ XNUMXB) แต่การกำหนดและการแสดงภาพ congeners ทั้งหมดยังคงใช้เวลานานและใช้งานง่ายไม่ดี
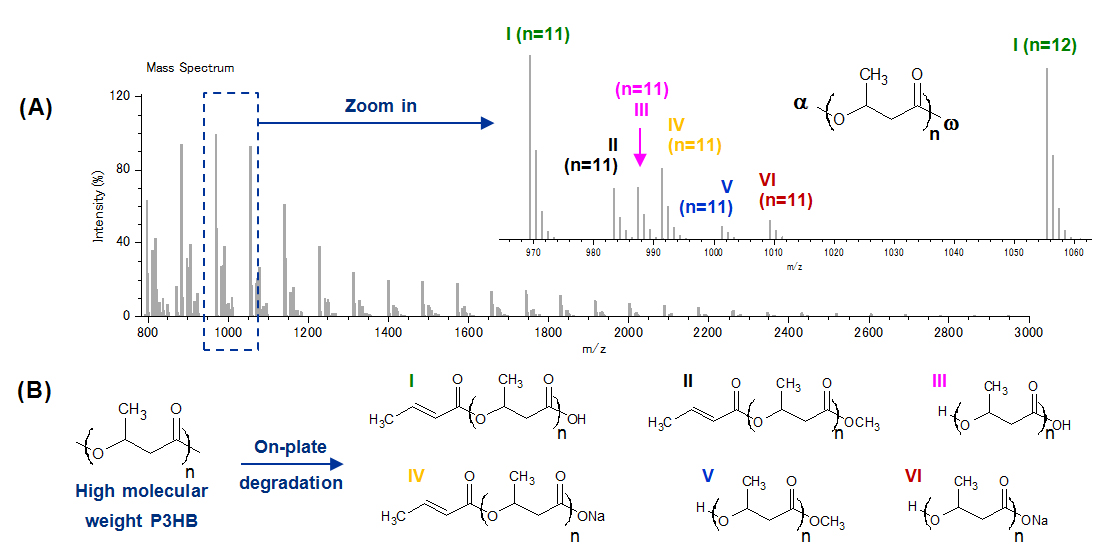
รูปที่ 1. (A) สเปกตรัมมวลของ MALDI SpiralTOF ของ P3HB ที่มีน้ำหนักโมเลกุลสูงหลังจากการสลายตัวบนจาน (สิ่งที่ใส่เข้าไป: ซูมช็อตด้วยการกำหนด) (B) ชุดไอออน P3HB ระบุ I-VI
แปลง KMD ปกติและปรับปรุงความละเอียด
พล็อต KMD ปกติที่คำนวณจากสเปกตรัมมวลของ MALDI จะแสดงกลุ่มเมฆของจุดที่เรียงตามแนวนอน (รูปที่ 2A). การซูมเข้าเป็นสิ่งจำเป็นเนื่องจากจุดต่างๆ ถูกจัดกลุ่มอยู่ภายในส่วนที่จำกัดของพล็อตเนื่องจากการเปลี่ยนแปลงที่จำกัดมากของ KMD กับการแปรผันของไอโซโทปหรือองค์ประกอบกลุ่มสุดท้าย [4] เห็นกลุ่มจุดที่แก้ไขได้ไม่ดีสองกลุ่ม (รูปที่ 2B) ในขณะที่คาดว่าจะมีหกซีรี่ส์เมื่อพิจารณาจากข้อมูลสเปกตรัม
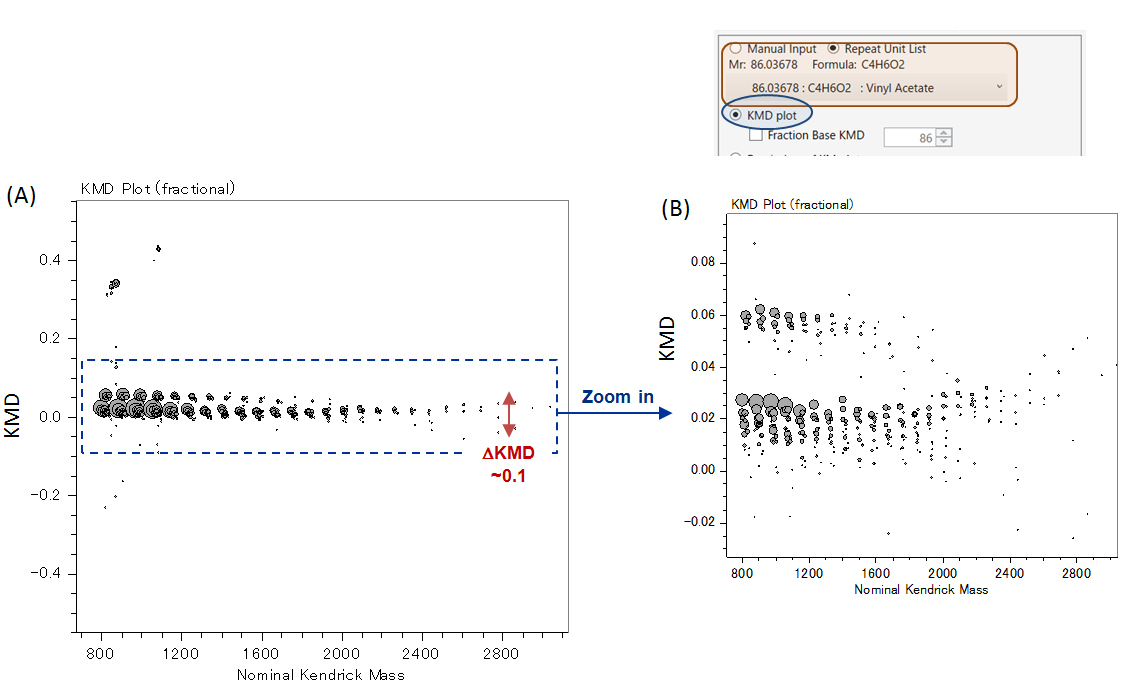
รูปที่ 2 พล็อต KMD ปกติแบบเต็มสเกลจากสเปกตรัมมวลของ P3HB กับ C4H6O2 เป็นหน่วยพื้นฐานโดยใช้ msRepeatFinder
การเปิดใช้งานตัวเลือก "Fraction Base KMD" ใน msRepeatFinder และลดค่าของ X จาก 86 (พล็อต KMD ปกติ) เป็น 85 พล็อต KMD ที่ปรับปรุงความละเอียดจะแยกซีรีส์ P3HB หกชุดออกจากกันอย่างมากจาก KMD ที่มีความหลากหลายมากขึ้นโดย เปลี่ยนองค์ประกอบ (รูปที่ 3A). การลด X ของหนึ่งหน่วยที่ X=84 พล็อต KMD ที่ปรับปรุงความละเอียดจะแก้ไขรูปแบบไอโซโทปเพิ่มเติม (1st สาย: 12ค, 2nd สาย: 13C1, …) และแยกแยะซีรีส์ทั้งหกได้ดียิ่งขึ้น (รูปที่ 3B, X=84)
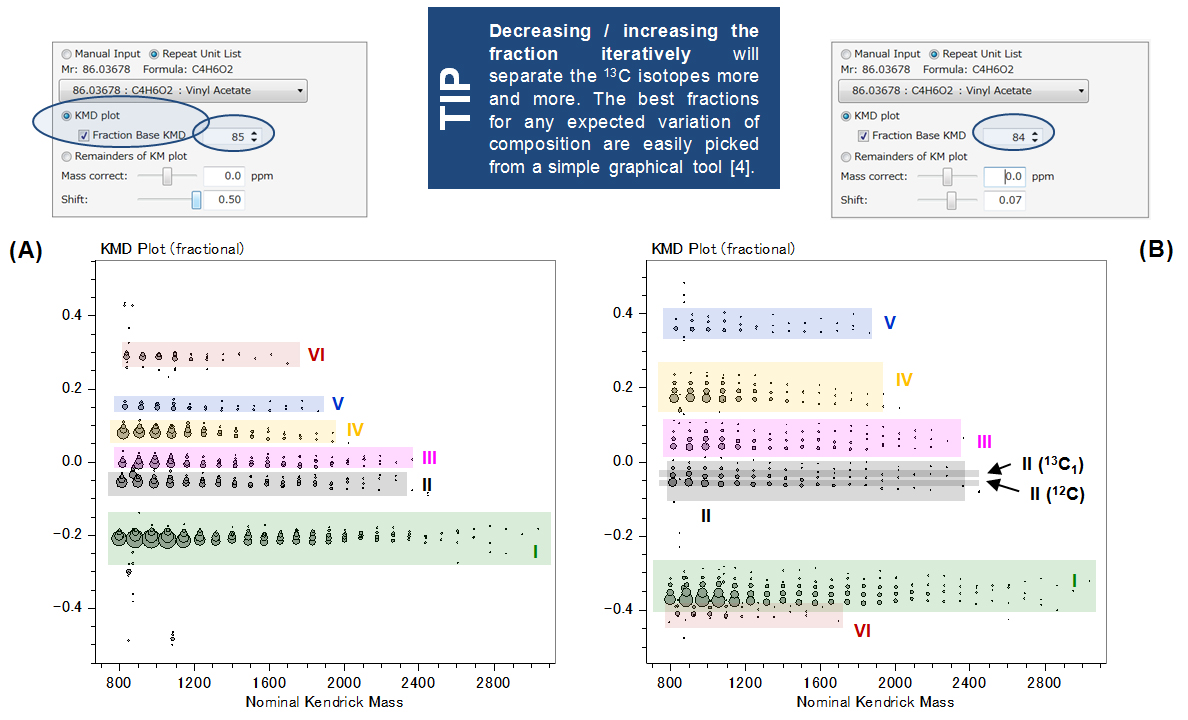
รูปที่ 3 แปลง KMD ฐานเศษส่วนแบบเต็มสเกลที่คำนวณด้วยหน่วยฐานเศษส่วน (A) 3HB/85 และ (B) 3HB/84 โดยใช้ msRepeatFinder
โหมดการจัดกลุ่มด้วยพล็อต KMD ที่ปรับปรุงความละเอียด
"โหมดการจัดกลุ่ม" ใน msRepeatFinder เมื่อรวมกับการวิเคราะห์ KMD ฐานเศษส่วนทำให้สามารถระบุชุดไอออนแต่ละชุดในสเปกตรัมมวลและพล็อตด้วยรหัสสีที่มองเห็นได้ผ่านการเลือกจุดอย่างง่าย (มะเดื่อ. 4). กำลังการแยกสูงที่เข้าถึงได้ด้วย 3HB/84 ช่วยให้สามารถเลือกพีคที่มีไอโซโทปเดียว (12C, จุดสีเขียวอ่อน) จากชุดหลัก I (จุดสีเขียวเข้ม) นอกจากนี้ยังสามารถซ่อนชุดไอออนที่ไม่ได้เลือกเพื่อช่วยเพิ่มเติมในการแสดงภาพชุดพอลิเมอร์จากจุดสูงสุดในพื้นหลัง
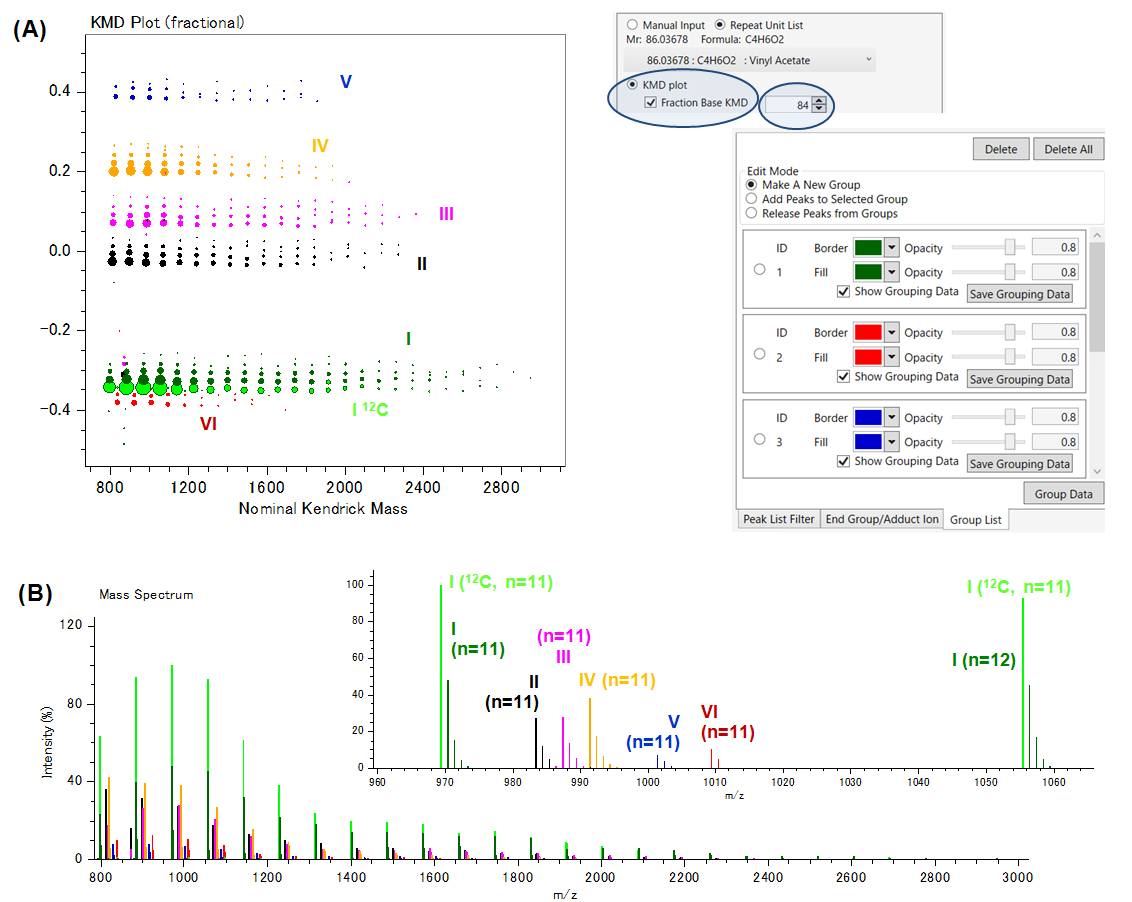
รูปที่ 4 (A) พล็อต KMD ฐานเศษส่วนคำนวณด้วยหน่วยฐานเศษส่วน 3HB/84 ในโหมดการจัดกลุ่มเพื่อติดฉลากอนุกรมในสเปกตรัมมวล (B)
สรุป
การวิเคราะห์ KMD กลายเป็นไดนามิกด้วยการเปิดตัว "Fraction Base KMD" ที่อาศัยวิธีการปรับปรุงความละเอียด เป็นเครื่องมือที่มีความอเนกประสงค์สูงเนื่องจากการลดหรือเพิ่มค่าของเศษส่วนจะนำไปสู่การแยกชุดไอออนที่น่าพอใจซึ่งแตกต่างกันไปตามองค์ประกอบของไอโซโทป กลุ่มสุดท้าย หรือไอออนเสริม สามารถปรับให้เข้ากับโฮโมโพลิเมอร์ประเภทใดก็ได้และข้อมูลแมสสเปกตรัมประเภทใดก็ได้ที่มีความแม่นยำปานกลางถึงสูงเป็นพิเศษ
กิตติกรรมประกาศ
บันทึกการใช้งานนี้เขียนขึ้นจากผลของโครงการวิจัยร่วมกับ Sayaka Nakamura, Thierry Fouquet และ Hiroaki Sato จากสถาบันวิจัยเพื่อเคมีที่ยั่งยืน สถาบันวิทยาศาสตร์และเทคโนโลยีอุตสาหกรรมขั้นสูงแห่งชาติ (AIST)
อ้างอิง
[1] S. Nakamura, T. Fouquet, H. Sato แยม. ซ. มวลสาร 2019, 30, 355.
[2] H. Sato, S. Nakamura, K. Teramoto, T. Sato แยม. สังคม แมสสเปกตรัม. 2014, 25, 1346
[3] ต. ฟูเก้, เอช. ซาโต้ ก้น เคมี. 2017, 89, 2682
[4] S. Nakamura, RB Cody, H. Sato, T. Fouquet เคมีก้น. 2019, 91, 2004
- หากต้องการดูฉบับพิมพ์ โปรดคลิกไฟล์ PDF นี้

PDF845KMB
สินค้าที่เกี่ยวข้อง
คุณเป็นผู้เชี่ยวชาญทางการแพทย์หรือบุคลากรที่เกี่ยวข้องกับการรักษาพยาบาลหรือไม่?
ไม่
โปรดทราบว่าหน้าเหล่านี้ไม่ได้มีวัตถุประสงค์เพื่อให้ข้อมูลเกี่ยวกับผลิตภัณฑ์แก่ประชาชนทั่วไป
