การแปลง KMD ที่เป็นฐานเศษส่วนสำหรับโคโพลีเอสเทอร์โพลี (3-ไฮดรอกซีบิวทีเรต-โค-3-ไฮดรอกซีวาเลอเรต) ที่มีน้ำหนักโมเลกุลสูง หลังจากการย่อยสลายด้วยอัลคาไลน์บนเพลตและการวิเคราะห์ SpiralTOF™ [แอปพลิเคชัน MALDI]
MStips ฉบับที่ 284
บทนำ
โพลิเมอร์ที่มีน้ำหนักโมเลกุลสูงมักเป็น MS-silent เนื่องจากมีการกระจายตัวสูงโดยธรรมชาติ (ĐM) หรือตรวจจับได้ในช่วงมวลสูงที่มีกำลังแยกต่ำ แมสสเปกโตรเมทรีความละเอียดสูง (HRMS) นั้นจำกัดอยู่ที่ช่วงมวลต่ำ (< 3000 Da) สำหรับการประเมินธรรมชาติของหน่วยที่ทำซ้ำและ/หรือกลุ่มสุดท้ายหรือการแยกสารประกอบไอโซบาริกอย่างชัดเจน ดังนั้น การย่อยสลายด้วยด่างแบบ "บนจาน" จึงได้รับการพัฒนาเป็นตัวอย่างการปรับสภาพล่วงหน้าบนเป้าหมายของ MALDI ด้วยโพลิเมอร์ 1 ใน XNUMX ของ ng เพื่อตัดโซ่โพลีเอสเตอร์อุตสาหกรรมขนาดยาวให้เป็นโอลิโกเมอร์สั้นที่คล้อยตาม MALDI-HRMS [XNUMX] ความซับซ้อนของแมสสเปกตรัมที่เกี่ยวข้องสามารถลดลงได้อย่างมากด้วยการวิเคราะห์ Kendrick Mass Defect (KMD) ที่ปรับปรุงความละเอียดที่เหมาะสมโดยใช้ตัวเลือก "ฐานเศษส่วน" ของ msRepeatFinder เพื่อสร้างแผนที่องค์ประกอบ
การทดลอง
โพลี(3-ไฮดรอกซีบิวทิเรต-โค-3-ไฮดรอกซีวาเลอเรต) (PHBV, 12 โมล% 3HV, Mn=1.4×105 กรัม โมล-1, ดM=2.5) มาจาก Sigma-Aldrich (เซนต์หลุยส์, มิสซูรี่, สหรัฐอเมริกา) ในขณะที่สารเคมีและตัวทำละลายอื่นๆ ทั้งหมดมาจาก FUJIFILM Wako Pure Chemical Corporation เว้นแต่จะระบุไว้เป็นอย่างอื่น PHBV 1 ไมโครลิตร (1 มก. มล -1 ใน THF) ถูกโยนทิ้งบนแผ่นเป้าหมาย MALDI แบบใช้แล้วทิ้ง (Hudson Surface Technology, NJ, USA) โซเดียมไฮดรอกไซด์ 1 ไมโครลิตรในเมทานอล-d4 ที่ 10 มก. มล-1 ถูกสะสมเพิ่มเติมและปล่อยให้แห้ง น้ำยาอัลคาไลน์ส่วนเกินถูกล้างด้วยน้ำกลั่น 1 μL ของ 2,4,6-trihydroxyaceto-phenone (Protea Biosciences, WV, USA) ใน THF ที่ 20 mg mL -1 ถูกนำไปใช้กับจุดตัวอย่าง แมสสเปกตรัมถูกบันทึกด้วยแมสสเปกโตรมิเตอร์ JMS-S3000 SpiralTOF™ แปลงคำนวณโดยใช้ msRepeatFinder 3.0 จากรายการสูงสุดที่ส่งออกโดยใช้ msTornado™
การวิเคราะห์ MS และ KMD ปกติ
การสลายตัวของ PHBV บนจานที่เป็นด่างจะปล่อยชุดโคโพลีเอสเทอร์แบบสั้นสูงสุดหกชุดที่ตรวจพบได้ง่ายใน MALDI-MS [1] (รูปที่ 1A). ด้วยการเปลี่ยนแปลงของ end-groups, 3HB และ 3HV เนื้อหาภายในหนึ่งชุดและรูปแบบไอโซโทปที่กำหนด การกำหนดจุดสูงสุดนั้นไม่ใช่เรื่องเล็กน้อยทั้งจากสเปกตรัมมวลหรือโครงร่าง KMD ปกติ [2] เนื่องจากชุดข้อมูลเหลื่อมกัน (รูปที่ 1B).
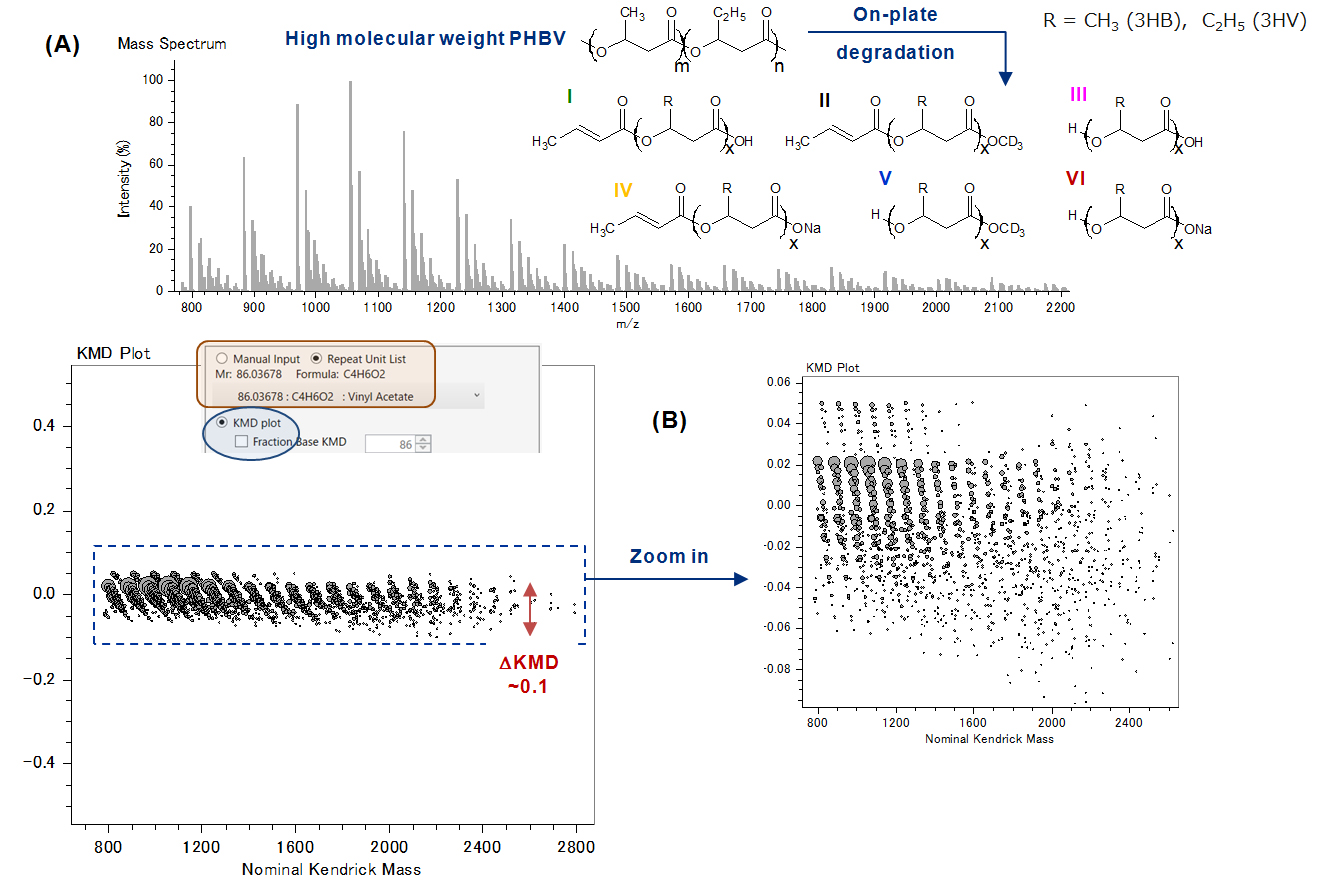
รูปที่ 1. (A) สเปกตรัมมวล MALDI spiralTOF™ ของ PHBV ที่มีน้ำหนักโมเลกุลสูงหลังจากการสลายตัวบนจาน (สิ่งที่ใส่เข้าไป: ชุดไอออนหลักที่ตรวจพบ I-VI) (B) พล็อต KMD ปกติเต็มรูปแบบ (ฐาน: C4H6O2, 3HB)
การวิเคราะห์ KMD ที่ปรับปรุงความละเอียดแบบ "ภาพเดียว"
ตรงกันข้ามกับพล็อต KMD ปกติแบบย่อ ชุดไอออนที่แตกต่างกันครอบคลุมช่วงทั้งหมดของ KMD (ตั้งแต่ -0.5 ถึง 0.5) ในพล็อต KMD ฐานเศษส่วนซึ่งคำนวณด้วย 3HB/135 (มะเดื่อ. 2) [3]. ภายในคลัสเตอร์หนึ่ง จุดจะแตกต่างกันตามเนื้อหาใน 3HB (การจัดแนวแนวนอน หน่วยฐาน) และใน 3HV (การวางแนวเฉียง) ในขณะที่องค์ประกอบของไอโซโทปจะแตกต่างจากคลัสเตอร์หนึ่งไปยังอีกกลุ่มหนึ่ง (12C หรือ 13Cx, มะเดื่อ. 2 พร้อมรหัสสี) พีคทั้งหมดที่ตรวจพบในสเปกตรัมมวลจึงถูกแยกหรือจัดกลุ่มอย่างได้เปรียบเพื่อการมอบหมายที่รวดเร็ว
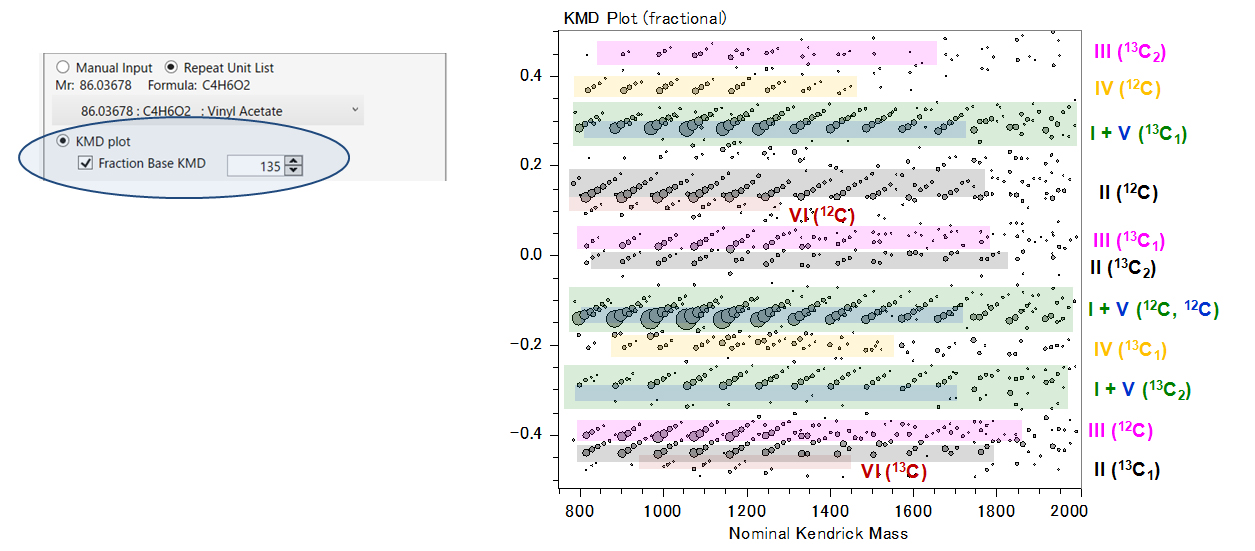
รูปที่ 2. พล็อต KMD "ฐานเศษส่วน" ที่มีหน่วยฐานเศษส่วน 3HB/135 โดยใช้ msRepeatFinder
การวิเคราะห์ KMD ที่ปรับปรุงความละเอียด "ตามลำดับ" [4]
ชุด I และ V เกือบจะซ้อนทับกันในพล็อต KMD ฐานเศษส่วนก่อนหน้า (มะเดื่อ. 2) ป้องกันการแยกตัว/โดดเดี่ยว เพื่อหลีกเลี่ยงปัญหานี้ คลัสเตอร์ monoisotopic จะถูกแยกออก (โหมดการจัดกลุ่ม รูปที่ 3A) ในขณะที่จุดอื่น ๆ ถูกซ่อนไว้เพื่อปรับปรุงการแสดงภาพ (รูปที่ 3B). ชุดไอออนโมโนไอโซโทปสองชุดถูกแยกออกโดยการเปลี่ยนเศษส่วน (3HB/117, รูปที่ 3C). จุดทั้งหมดถูกแยกออกจากกันอย่างสมบูรณ์ เป็นโค-โอลิโกเมอร์ชนิดโมโนไอโซโทป (12C) ที่มีเนื้อหาแตกต่างกันใน 3HB (การจัดแนวในแนวนอน) และ 3HV (การจัดแนวเฉียง โครงสร้างในส่วนแทรก)
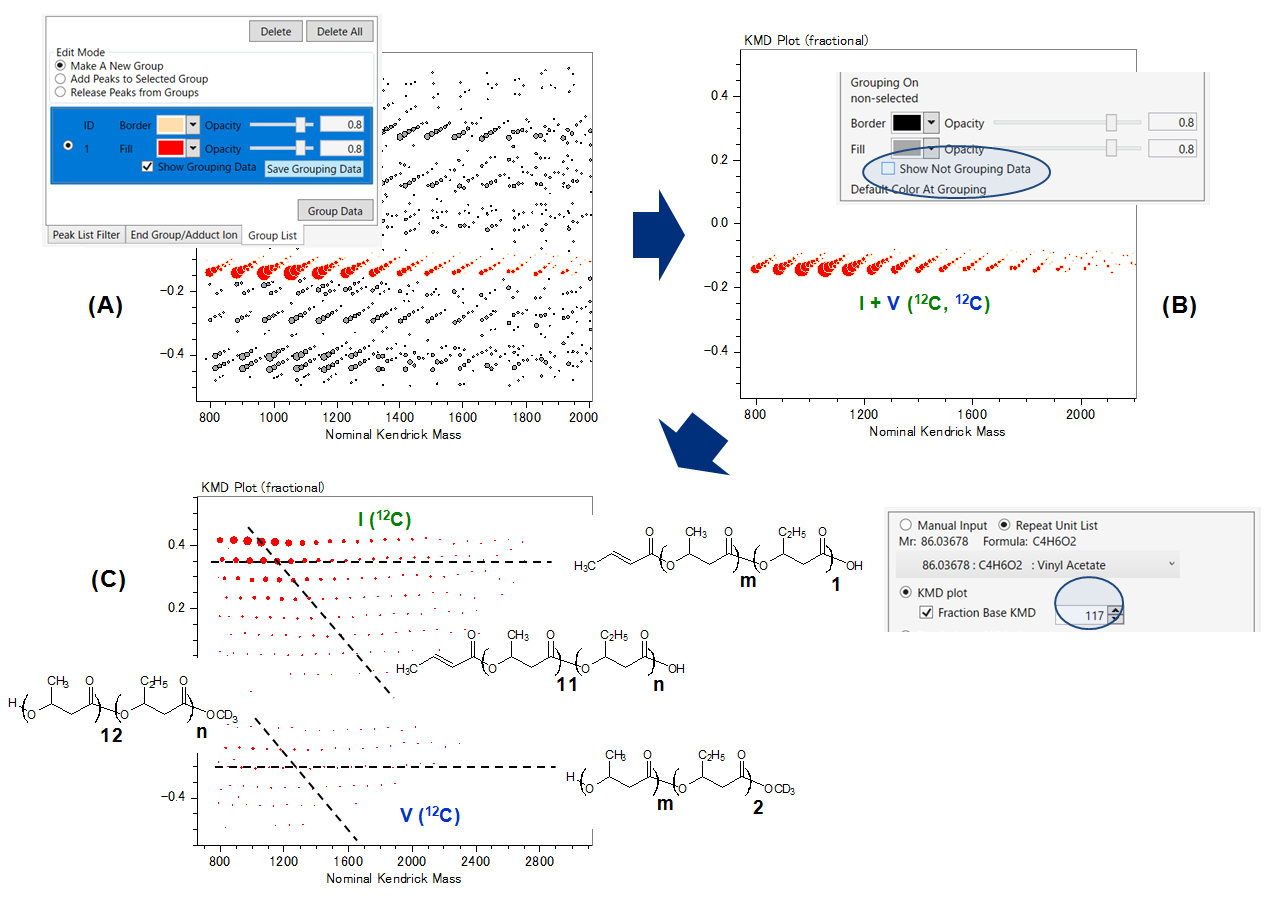
รูปที่ 3. การวิเคราะห์ KMD "ฐานเศษส่วน" ตามลำดับโดยใช้ msRepeatFinder
การประมวลผลข้อมูลทางเลือก: สเปกตรัมมวลที่แยกไอโซโทป (msTornado™)
การใช้ชุดข้อมูลทั้งหมดช่วยลดความเสี่ยงของชุดไอออนย่อยที่ขาดหายไป แต่ไอโซโทปทำให้การประมวลผลข้อมูลซับซ้อนขึ้นด้วยคลัสเตอร์หลายชุดสำหรับชุดโคพอลิเมอร์ที่กำหนด (มะเดื่อ. 2). สเปกตรัมมวลอาจถูกแยกไอโซโทปโดยอัตโนมัติแทนโดยใช้ msTornado™ เพื่อทำให้การแสดงภาพง่ายขึ้น อย่างไรก็ตาม แม้จะมีการแยกไอโซโทปออกแล้ว แผนภาพ KMD ปกติยังคงล้มเหลวในการแยกอนุกรมไอออน (รูปที่ 4A). แทน พล็อต KMD ฐานเศษส่วนจะคำนวณด้วยหน่วยเศษส่วนอื่น 3HB/92 (รูปที่ 4B) แยกชุดโคพอลิเมอริกไอออนทั้งหกชุดออกจากกันอย่างมากในแผนภาพเดียว ทำให้สามารถเลือก/กำหนดโค-โอลิโกเมอร์ได้อย่างรวดเร็วด้วยการมองเห็นที่ไม่มีใครเทียบได้ [1] ฐานเศษส่วนแตกต่างจากกรณีก่อนหน้า (135 หรือ 117 ถึง 92) เนื่องจากความจำเป็นในการแยกแตกต่างกัน (เนื้อหาใน 3HB, 3HV และกลุ่มสุดท้ายเท่านั้น ไม่มีไอโซโทป) [5]
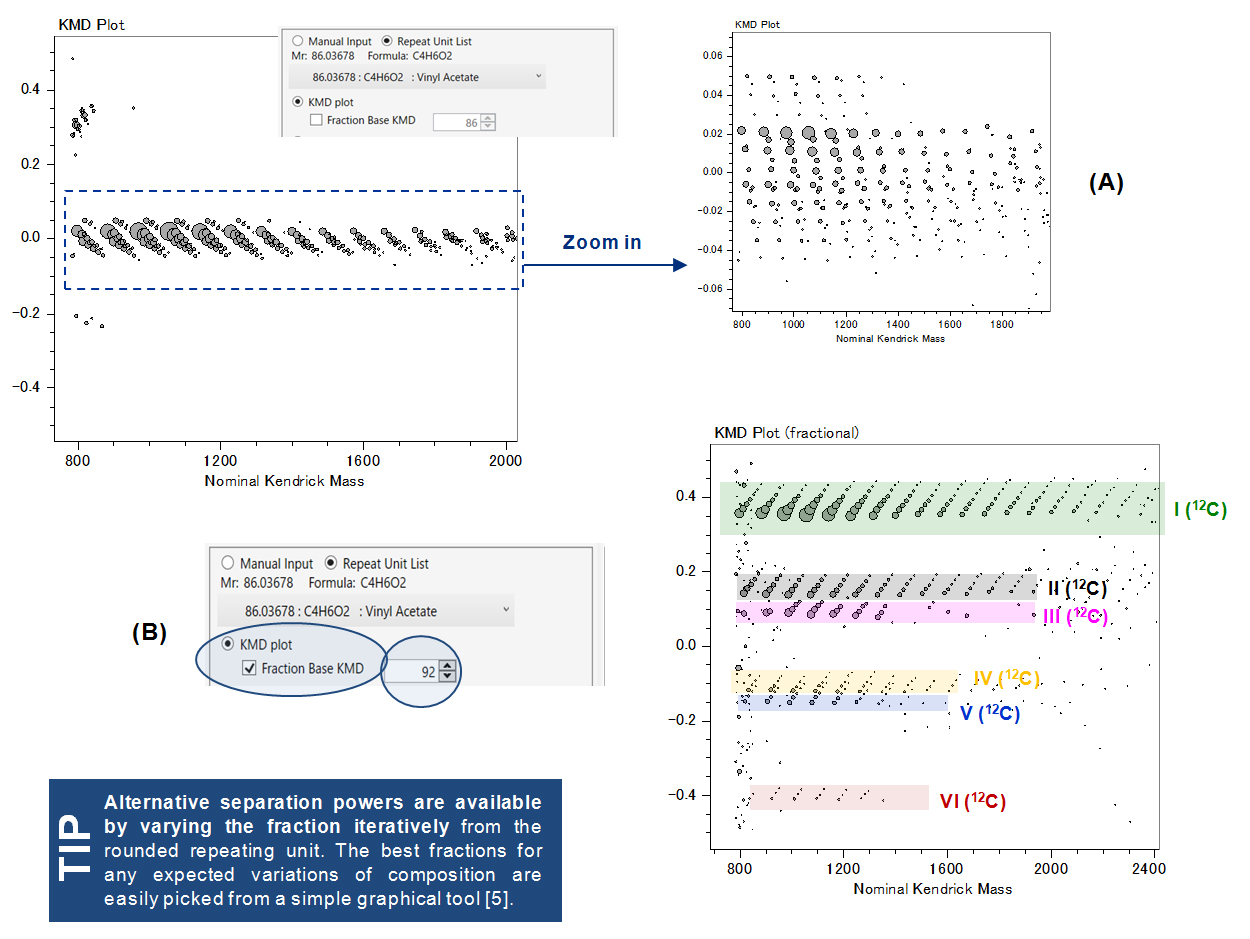
รูปที่ 4. (A) ปกติและ (B) พล็อต KMD ฐานเศษส่วน (3HB/92) จากสเปกตรัมมวลแบบแยกไอโซโทปโดยใช้ msRepeatFinder
สรุป
แผนภาพ KMD ที่แตกต่างกันจะคำนวณจากสเปกตรัมมวลเดียวกันโดยการเปลี่ยนแปลงเศษส่วนเพื่อสร้างแผนที่องค์ประกอบที่เหมาะสม ขึ้นอยู่กับความต้องการในการแสดงภาพ (เช่น การแยกโดย 13ไอโซโทป C, กลุ่มสุดท้าย, ปริมาณโคโมโนเมอริก หรือการรวมกันของพวกมัน) การใช้การย่อยสลายด่างบนจาน (การเตรียมตัวอย่างล่วงหน้าสำหรับตัวอย่างที่มีน้ำหนักโมเลกุลสูง) และความสามารถในการกรอง msRepeatFinder (การประมวลผลข้อมูล) การวิเคราะห์ "ฐานเศษส่วน KMD" เป็นเครื่องมืออเนกประสงค์ที่เหมาะสำหรับชุดข้อมูลโคพอลิเมอร์จำนวนมาก
กิตติกรรมประกาศ
บันทึกการใช้งานนี้เขียนขึ้นจากผลของโครงการวิจัยร่วมกับ Sayaka Nakamura, Thierry Fouquet และ Hiroaki Sato จากสถาบันวิจัยเพื่อเคมีที่ยั่งยืน สถาบันวิทยาศาสตร์และเทคโนโลยีอุตสาหกรรมขั้นสูงแห่งชาติ (AIST)
อ้างอิง
[1] S. Nakamura, T. Fouquet, H. Sato แยม. สังคม แมสสเปกตรัม. 2019, 30, 355
[2] H. Sato, S. Nakamura, K. Teramoto, T. Sato แยม. สังคม แมสสเปกตรัม. 2014, 25, 1346
[3] ต. ฟูเก้, เอช. ซาโต้ ทางทวารหนัก Chem. 2017, 89, 2682
[4] Q. Zheng, M. Morimoto, H. Sato, T. Fouquet เชื้อเพลิง 2019, 235, 944
[5] S. Nakamura, RB Cody, H. Sato, T. Fouquet ก้นเคมี- 10.1021/acs.analchem.8b04371
- โปรดดูไฟล์ PDF สำหรับข้อมูลเพิ่มเติม
หน้าต่างอื่นจะเปิดขึ้นเมื่อคุณคลิก 
PDF768KMB
สินค้าที่เกี่ยวข้อง
คุณเป็นผู้เชี่ยวชาญทางการแพทย์หรือบุคลากรที่เกี่ยวข้องกับการรักษาพยาบาลหรือไม่?
ไม่
โปรดทราบว่าหน้าเหล่านี้ไม่ได้มีวัตถุประสงค์เพื่อให้ข้อมูลเกี่ยวกับผลิตภัณฑ์แก่ประชาชนทั่วไป
