การเปรียบเทียบวิธีการสร้างภาพ 3 มิติในกล้องจุลทรรศน์อิเล็กตรอนสำหรับวัสดุชีวภาพ
JEOL NEWS ฉบับที่ 53 No.8
โทโมฮิโระ ฮารุตะ
ฝ่ายจัดการแอปพลิเคชัน JEOL Ltd.
TEM tomography ถูกนำมาใช้กันอย่างแพร่หลายสำหรับการวิเคราะห์สามมิติ (3D) ของโครงสร้างที่ดี เมื่อเร็วๆ นี้ วิธีการวิเคราะห์โครงสร้าง 3 มิติแบบใหม่ด้วย SEM, FIB-SEM, Serial Block-Face SEM (SBF-SEM) และการตรวจเอกซเรย์แบบอาร์เรย์ ได้รับความสนใจจากนักวิจัยจำนวนมากเนื่องจากการปรับปรุงประสิทธิภาพของคอมพิวเตอร์และ SEM วิธีการเหล่านี้เหมาะสำหรับตัวอย่างทางชีววิทยาเนื่องจากพื้นที่ที่สังเกตได้กว้างมากเมื่อเปรียบเทียบกับการตรวจเอกซเรย์ TEM แต่ละวิธีมีข้อดีและข้อเสียของตัวเอง อย่างไรก็ตาม มีตัวอย่างการเปรียบเทียบระหว่างวิธีการเหล่านี้เพียงเล็กน้อยโดยใช้ตัวอย่างเดียวกัน ในการศึกษานี้ เราสร้างโครงสร้าง 3 มิติของตัวอย่างเดียวกัน (E.coli และฟาจ) โดย FIB-SEM, วิธี SBF-SEM, Array tomography และ TEM tomography และตรวจสอบข้อดีและข้อเสียของวิธีการเหล่านี้
บทนำ
สิ่งสำคัญคือต้องรู้โครงสร้าง 3 มิติและการแปลของออร์แกเนลล์และโปรตีนเชิงซ้อนที่เป็นส่วนประกอบของเซลล์เพื่อทำความเข้าใจหน้าที่ของเซลล์และเนื้อเยื่อ ข้อมูลนี้มีบทบาทสำคัญไม่เพียงแต่ในการวิจัยเชิงวิชาการเท่านั้นแต่ยังรวมถึงการพัฒนาการบำบัดโรคร้ายแรงอีกด้วย ก่อนหน้านี้ การตรวจเอกซเรย์ TEM ถูกใช้สำหรับการสังเกตโครงสร้าง 3 มิติของโครงสร้างนาโนเหล่านี้ ในการตรวจเอกซเรย์ TEM เรานำไมโครกราฟของภาพที่เอียงแบบอนุกรม และสร้างภาพ 3 มิติขึ้นใหม่โดยการฉายภาพด้านหลังชุดภาพที่ได้ เอกซ์เรย์ TEM ให้ภาพ 3 มิติที่มีความละเอียดสูง และยังมีข้อจำกัดด้านความหนาที่กำหนดโดยการส่งผ่านลำอิเล็กตรอน ดังนั้นจึงเป็นเรื่องยากที่จะสังเกตภาพ 3 มิติของทั้งเซลล์หรือเนื้อเยื่อโดย TEM tomography จุลชีววิทยาหรือชีววิทยาของเซลล์เมื่อเร็วๆ นี้มีความเกี่ยวข้องอย่างยิ่งกับอณูชีววิทยา ดังนั้นจึงเป็นสิ่งสำคัญที่จะเปิดเผยอิทธิพลของสัณฐานวิทยาและการกระจายของโครงสร้างนาโนบนเนื้อเยื่อและทั้งเซลล์ ด้วยเหตุผลดังกล่าว จึงมีการพัฒนาวิธีการใหม่ด้วยกล้องจุลทรรศน์ ซึ่งช่วยให้เราสังเกตพื้นที่ขนาดใหญ่ที่รู้จักทั้งเซลล์หรือเนื้อเยื่อ และสามารถรักษาความละเอียดสูงได้ในขณะเดียวกันก็จดจำออร์แกเนลล์และโปรตีนเชิงซ้อน ในการศึกษานี้ เรามุ่งเน้นไปที่วิธีการสังเกต 3D ใหม่สามวิธี ได้แก่ FIB-SEM, SBF-SEM และ Array tomography [1, 2, 3] (รูปที่ 1)
รูปที่ 1 แผนผังของวิธีการสร้างใหม่ 3 มิติ
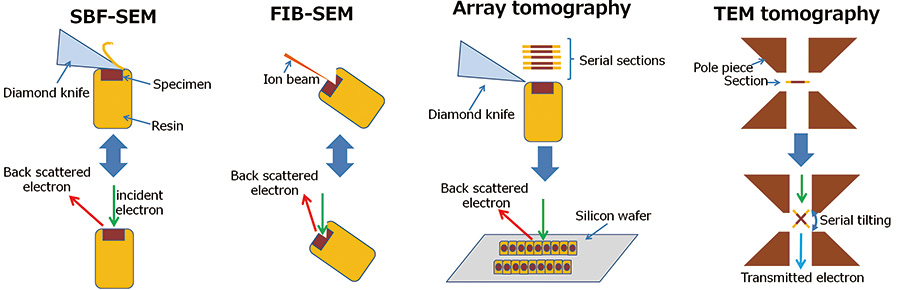
- SBF-SEM:
SBF-SEM ทำซ้ำการตัดตัวอย่างด้วยมีดเพชรและการสังเกตภาพอิเล็กตรอนที่กระจัดกระจายของพื้นผิวตัวอย่างใหม่ในห้องตัวอย่าง - FIB-SEM:
FIB-SEM ทำซ้ำการสปัตเตอร์ของตัวอย่างด้วยลำแสงแกลเลียมไอออนที่โฟกัสและการสังเกตภาพอิเล็กตรอนที่กระจัดกระจายของพื้นผิวตัวอย่างใหม่ในห้องตัวอย่าง - เอกซเรย์อาร์เรย์:
ส่วนแบบอนุกรมเตรียมด้วย ultramicrotome และติดตั้งบนพื้นผิวที่แข็ง เช่น แผ่นเวเฟอร์ซิลิกอน ส่วนอนุกรมเหล่านี้ใช้สำหรับภาพอิเล็กตรอนที่กระจัดกระจายทีละภาพและเรียงซ้อนกันตามลำดับ - เอกซเรย์ TEM:
การตรวจเอกซเรย์ TEM ใช้ภาพฉายภาพแบบเอียงต่อเนื่องโดย TEM และสร้างโครงสร้าง 3 มิติภายในขึ้นใหม่โดยการฉายภาพด้านหลัง
FIB-SEM คือ SEM ที่ติดตั้งคอลัมน์ Focused ion beam (FIB) ตัวอย่างในห้องตัวอย่างถูกสปัตเตอร์โดย FIB และ SEM สามารถสังเกตพื้นผิวที่สปัตเตอร์ได้ FIB ไม่เป็นที่นิยมในด้านชีววิทยา แต่เป็นที่นิยมในด้านวัสดุศาสตร์ เนื่องจากตัวอย่างในวัสดุศาสตร์ (โลหะ เซรามิก ฯลฯ) ยากเกินกว่าที่จะเฉือนด้วยมีดเพชร แต่สามารถทำให้เป็นส่วนบางหรือเปิดผิวใหม่ได้ด้วยการสปัตเตอร์ด้วย FIB ในการสังเกตโครงสร้าง 3 มิติด้วย FIB-SEM เราจะพ่นพื้นผิวของชิ้นงานทดสอบ และสังเกตพื้นผิวที่เปิดใหม่โดยการถ่ายภาพอิเล็กตรอนแบบกระจัดกระจายกลับด้วย SEM และทำซ้ำขั้นตอนการสปัตเตอร์และการสังเกต สุดท้าย เราสามารถสร้างโครงสร้าง 3D ขึ้นใหม่ได้โดยการซ้อนภาพของชุดส่วนอนุกรม [1] ข้อดีของ FIB-SEM คือการกำหนดตำแหน่งสปัตเตอร์ความแม่นยำสูงและการเตรียมชิ้นส่วนบางจากตัวอย่างแข็ง เช่น กระดูกและโลหะ ในทางกลับกัน ข้อเสียคือพื้นที่การสังเกตที่แคบและความเร็วในการสปัตเตอร์ช้า
ใน SBF-SEM เราใช้ SEM ที่มีกลไกในการตัดตัวอย่างด้วยมีดเพชรในห้องตัวอย่าง ในการสร้างภาพ 3 มิติขึ้นใหม่ พื้นผิวของตัวอย่างจะถูกหั่นด้วยมีดเพชรในห้องตัวอย่าง และ SEM จะสังเกตเห็นพื้นผิวใหม่ที่เปิดเผย ภาพ 3 มิติถูกสร้างขึ้นใหม่โดยการซ้อนภาพของชุดส่วนอนุกรม เช่น FIB-SEM [2] ข้อดีของ SBF-SEM คือความเร็วตัดสูงและมีพื้นที่ตัดกว้าง เนื่องจากตัวอย่างจะถูกตัดด้วยมีดเพชร ในทางกลับกัน มีข้อเสียตรงที่ SBF-SEM ต้องการการเตรียมตัวอย่างพิเศษ (เช่น วิธี NCMIR) เนื่องจากตัวอย่างจะต้องเตรียมด้วยค่าการนำไฟฟ้าสูงและการย้อมสีโลหะหนักอย่างแรง [3] คุณภาพการถนอมอาหารของโครงสร้างนาโนโดยการเตรียมตัวอย่างพิเศษนี้ด้อยกว่าการเตรียมแบบธรรมดาสำหรับ TEM แม้ว่าค่าการนำไฟฟ้าของตัวอย่างจะเพิ่มขึ้นโดยการเตรียมตัวอย่างพิเศษ แต่บางครั้งอาจมีประจุตัวอย่าง
ในการตรวจเอกซเรย์แบบอาร์เรย์ ขั้นแรกเราจะเตรียมตัวอย่างริบบ้อนส่วนที่บางเฉียบโดยใช้ ultramicrotome และติดตั้งส่วนอนุกรมบนเวเฟอร์ที่เป็นสื่อกระแสไฟฟ้าเช่นเวเฟอร์ซิลิกอน ส่วนอนุกรมเหล่านี้ได้รับการสังเกตทีละส่วนด้วย SEM และภาพ 3 มิติจะถูกสร้างขึ้นใหม่เพื่อซ้อนภาพตามลำดับ [4] ข้อดีของวิธีนี้มีดังนี้ การปราบปรามการชาร์จตัวอย่างที่บางมากและติดตั้งบนเวเฟอร์นำไฟฟ้า ต้นทุนเริ่มต้นต่ำ เนื่องจากวิธีนี้ต้องการเพียง SEM ทั่วไปและ ultramicrotome เท่านั้น และเราสามารถใช้ตัวอย่างที่ดีที่เตรียมโดยเทคนิคการเตรียมแบบทั่วไปสำหรับ TEM ข้อเสียของวิธีนี้คือต้องใช้ความพยายามอย่างมาก เช่น การจัดเตรียมชุดส่วนอนุกรม
มีการอภิปรายเพียงเล็กน้อยเพื่อเปรียบเทียบคุณลักษณะและข้อมูลจำเพาะของวิธีการทั้งสามนี้ เนื่องจากหลักการของวิธีการสร้าง 3D ใหม่ด้วย SEM เกือบจะเหมือนกัน นั่นคือ สามวิธีช่วยให้เราสร้างส่วนต่อเนื่องและสังเกตส่วนใหม่ได้ ในการศึกษานี้ เราสังเกตโครงสร้าง 3 มิติของตัวอย่างเดียวกันด้วยวิธีการเหล่านี้ และเปรียบเทียบผลลัพธ์ สุดท้าย เราจะหารือและสรุปคุณสมบัติของวิธีการเหล่านี้ และตัวอย่างและการวิเคราะห์ใดที่เหมาะสม นอกจากนี้เรายังพยายามวิเคราะห์ตัวอย่างบางส่วนโดยใช้วิธีการที่เหมาะสม
วัสดุและวิธีการ
เราใช้ E.coli ติดเชื้อโดย T4 phage, λ phage และ φ174 phage เป็นตัวอย่าง Phages เป็นไวรัสชนิดหนึ่งที่ติดเชื้อแบคทีเรีย ขนาดไม่เกิน 200 นาโนเมตร และเป็นไปไม่ได้ที่จะสังเกตสัณฐานวิทยาของพวกมันโดยไม่ใช้กล้องจุลทรรศน์อิเล็กตรอน (รูปที่ 2 ac)
ฟาจฉีดจีโนมของพวกมัน เช่น DNA หรือ RNA เข้าไปใน E.coli เมื่อฟาจเกาะติด E.coli. ฟาจที่ติดเชื้อเข้าครอบงำระบบการถอดรหัส ระบบการแปล และระบบการจำลองแบบของเซลล์เจ้าบ้าน และเพิ่มจำนวนตัวเองในเซลล์เจ้าบ้าน ในขั้นตอนนี้ เราสามารถสังเกตการงอกของฟาจใน E.coli (รูปที่ 2 df) เมื่อฟาจแพร่กระจายอย่างเพียงพอในโฮสต์ ฟาจจะระเบิดและออกจากเซลล์โฮสต์ ฟาจที่แพร่ขยายใหม่จะเกาะติดและทำให้เซลล์โฮสต์ใหม่ติดเชื้อ
ขั้นแรก เราซ้อนวุ้น LB แบบนิ่มที่บรรจุ E.coli บนวุ้น LB แบบแข็ง Phages ติดเชื้อ E.coli โดยการวางฟาจที่มีสารละลายลงบนวุ้นนี้ เราสามารถระบุพื้นที่ที่ฟาจติดเชื้อได้ E.coli เป็นแผ่นโลหะหลังจากฟักตัวของวุ้นนี้และได้รับเชื้อฟาจ E.coli โดยเอาโล่เหล่านี้ออก
สำหรับ TEM tomography และ Array tomography ตัวอย่างถูกเติมล่วงหน้าโดย 2.5% กลูตารัลดีไฮด์ใน 0.1 โมลาร์โซเดียมคาโคดิเลต (pH 7.2) และหลังการแก้ไข 1% OsO4 ในโซเดียมคาโคดิเลต 0.1 โมลาร์ (pH 7.2) ตัวอย่างคงที่ถูกฝังในอีพอกซีเรซิน ตัวอย่างถูกหั่นด้วย ultramicrotome เป็นส่วนบาง ๆ ซึ่งติดตั้งบนแผ่นเวเฟอร์ซิลิกอนสำหรับการตรวจเอกซเรย์แบบอาร์เรย์ ส่วนบางเหล่านี้ถูกย้อมด้วยยูเรเนียมอะซิเตทและตะกั่วซิเตรต
สำหรับ FIB-SEM และ SBF-SEM เราใช้วิธี NCMIR สำหรับคราบโลหะหนักอย่างแรง [3] FIB-SEM ยังสามารถใช้ตัวอย่างที่จัดเตรียมโดยวิธีทั่วไป และในการศึกษานี้ เราใช้วิธี NCMIR เพื่อให้ได้ภาพที่มีความเปรียบต่างสูง หลังจากการตรึงและการย้อมสี ตัวอย่างถูกฝังในอีพอกซีเรซิน เงื่อนไขการสังเกตแสดงในตารางที่ 1
ภาพส่วนอนุกรมที่ได้รับจาก FIB-SEM, SBF-SEM และเอกซ์เรย์เอกซ์เรย์ถูกจัดเรียงโดยฟิจิ [5] และภาพ 3 มิติถูกสร้างขึ้นใหม่โดย stacker (System In Frontier Inc. Japan) ในการตรวจเอกซเรย์ TEM นั้น TEMography ถูกใช้เพื่อได้มาซึ่งภาพที่เอียงแบบอนุกรมและการสร้าง 3D ขึ้นใหม่ ภาพ 3 มิติที่ได้มานั้นถูกแบ่งส่วนและวิเคราะห์โดย Colorist (System In Frontier Inc. Japan)
รูปที่ 2 ภาพ TEM ของฟาจ
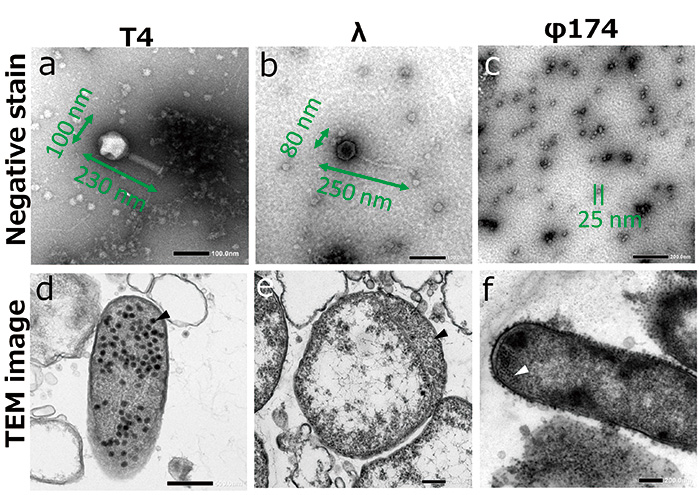
ภาพที่เปื้อนลบของฟาจ (ac) และการแพร่กระจายใน E.coli (df, หัวลูกศร).
ตารางที่ 1 เงื่อนไขการสังเกตของวิธีการสังเกต 3 มิติ
| SBF-SEM | FIB-SEM | เอกซเรย์อาร์เรย์ | TEM เอกซเรย์ | |
|---|---|---|---|---|
| ตราสาร | JSM-7800F พร้อม Gatan 3View 2XP | JIB-4610F | เจเอสเอ็ม-7800F | เจม-1400พลัส |
| พื้นที่การได้มา (X,Y) | 24 × 24 ไมโครเมตร2 | 18 × 24 ไมโครเมตร2 | 19 × 24 ไมโครเมตร2 | 3.5 × 3.5 ไมโครเมตร2 |
| ความลึกของการได้มา (Z) | 7.5 ไมครอน (50 นาโนเมตร × 150 ภาพ) | 10.8 ไมครอน (20 นาโนเมตร × 543 ภาพ) | 1.4 ไมครอน (70 นาโนเมตร × 20 ภาพ) | นาโนเมตร 200 |
| ขนาดพิกเซล (X,Y) | นาโนเมตร 3 | นาโนเมตร 9.4 | นาโนเมตร 4.7 | นาโนเมตร 1.72 |
| ขนาดพิกเซล (Z) | นาโนเมตร 50 | นาโนเมตร 20 | นาโนเมตร 70 | นาโนเมตร 1.72 |
| จำนวนพิกเซล | 8192 8192 × | 1920 2560 × | 3840 5120 × | 2048 2048 × |
| แรงดันไฟฟ้าเร่ง | 1.0 kV | 3.0 kV | 7.0 kV | 120 kV |
| เวลาที่ได้มา | 2.5 ชั่วโมง | 48 ชั่วโมง | 4 ชั่วโมง | 1 ชั่วโมง |
ผลลัพธ์และการอภิปราย
การเปรียบเทียบวิธีการสร้างใหม่ 3 มิติโดยใช้กล้องจุลทรรศน์อิเล็กตรอน
เราสังเกตภาพ 2 มิติโดยใช้ FIB-SEM, SBF-SEM (ภาพอิเล็กตรอนกระจัดกระจายของพื้นผิวของตัวอย่างจำนวนมาก แรงดันไฟฟ้าเร่งคือ 1.0 kV และ 3.0 kV ตามลำดับ) เอกซ์เรย์แบบอาร์เรย์ (ภาพอิเล็กตรอนที่กระจัดกระจายกลับของตัวอย่างบางส่วนที่ติดตั้งบนแผ่นเวเฟอร์ซิลิคอน แรงดันไฟเร่งเท่ากับ 7.0 kV) และ TEM tomography (ภาพฉายของตัวอย่างบางส่วน ไม่มีการเอียง แรงดันเร่งคือ 120 kV) FIB-SEM และ SBF-SEM สำหรับการสังเกตพื้นผิวของตัวอย่างจำนวนมากนั้นด้อยกว่า Array tomography และ TEM tomography สำหรับการสังเกตส่วนที่บางในความคมชัดของภาพ (รูปที่ 3) เนื่องจากในตัวอย่างนี้ พื้นที่สังเกตการณ์ส่วนใหญ่ใช้เรซินที่มีค่าการนำไฟฟ้าต่ำเท่านั้น และเราต้องสังเกตด้วยแรงดันไฟฟ้าที่เร่งความเร็วต่ำเพื่อระงับการชาร์จ คุณภาพของภาพเอกซเรย์แบบอาร์เรย์จะเท่ากับคุณภาพของภาพเอกซเรย์แบบ TEM ผลลัพธ์นี้ชี้ให้เห็นว่าการปรับปรุงการนำตัวอย่างและการสังเกตที่แรงดันไฟฟ้าเร่งสูงเป็นปัจจัยสำคัญในการสังเกตตัวอย่างทางชีววิทยาในการสังเกตโครงสร้าง 3 มิติด้วย SEM (รูปที่ 3 c) นอกจากนี้ คุณภาพของภาพ FIB-SEM และ SBF-SEM ยังต่ำเมื่อเทียบกับวิธีอื่นๆ อีก XNUMX วิธี แต่ยังคงความละเอียดในการจดจำอนุภาคฟาจไว้
ต่อไป เราเปรียบเทียบผลลัพธ์ของการสร้างใหม่ 3 มิติ ในการตรวจเอกซเรย์ TEM ความหนาของชิ้น (200 นาโนเมตร) มีขนาดเล็กกว่าเส้นผ่านศูนย์กลางของ E.coli (ประมาณ 2 ไมครอน) เพื่อให้ภาพทั้งหมดเป็นหนึ่ง E.coli ไม่สามารถสร้างใหม่เป็นแบบ 3 มิติได้ (รูปที่ 4 ง) ในทางกลับกัน SBF-SEM และ FIB-SEM ประสบความสำเร็จในการสังเกตสามมิติของปริมาณมาก (รูปที่ 3 a, b) ในการตรวจเอกซเรย์แบบอาร์เรย์ พื้นที่สังเกตของระนาบ XY นั้นเทียบได้กับพื้นที่ของวิธี SBF และ FIB-SEM อย่างไรก็ตาม ความหนาของปริมาตรการสังเกตลดลงเนื่องจากชิ้นที่เตรียมไว้มีเพียง 4 ชิ้นในการศึกษานี้ (รูปที่ 20 ค) แม้ว่าจะต้องใช้ทักษะที่พัฒนาเต็มที่และใช้เวลาในการเรียนรู้นาน แต่เราคิดว่ามันเป็นไปได้ที่จะเพิ่มความหนาของภาพการสร้างใหม่ 4 มิติโดยการเตรียมส่วนอนุกรมจำนวนมากและเพิ่มจำนวนไมโครกราฟที่ถ่าย ในการสกัดหนึ่ง E.coli และการแบ่งส่วนของฟาจที่มีอยู่ใน E.coli และเยื่อหุ้มเซลล์นอกเซลล์ของ E.coli เราสามารถรับรู้สัณฐานวิทยาของ E.coli และการกระจายเฟส 3 มิติโดยใช้วิธีการใดก็ได้ แม้ว่าจะมีความแตกต่างในความละเอียดเชิงพื้นที่ (รูปที่ 4 เอ๊ะ) เมื่อมุ่งเน้นไปที่อนุภาคฟาจเดียว รูปร่างของฟาจจะขยายออกไปในทิศทาง Z ใน SBF-SEM และการตรวจเอกซเรย์แบบอาร์เรย์ เนื่องจากความละเอียดตาม Z นั้นไม่เพียงพอต่อขนาดของฟาจ (รูปที่ 4 i, k) ใน FIB-SEM แม้ว่าฟาจจะถูกสร้างขึ้นใหม่เป็นรูปทรงลูกบอล อย่างไรก็ตาม รายละเอียดของภาพการสร้าง 3D ใหม่จะสูญหายไปเมื่อเทียบกับผลลัพธ์ของ TEM tomography (รูปที่ 4 j, l) เมื่อพิจารณาจากผลการเปรียบเทียบ พบว่า การวิเคราะห์เชิงปริมาณของสัณฐานวิทยาของ E.coli และการวิเคราะห์การกระจายของฟาจใน E.coli สามารถทำได้โดยวิธีการสร้างใหม่ 3 มิติด้วย SEM ในขณะเดียวกันการตรวจเอกซเรย์ TEM ให้ความละเอียดเชิงพื้นที่สูงซึ่งจำเป็นสำหรับการสังเกตโครงสร้างนาโนเช่นฟาจ ได้ผลลัพธ์ที่คล้ายคลึงกันมากกับ SBF-SEM และ FIB-SEM ซึ่งมีพื้นฐานทั่วไปในการหั่นและสังเกตตัวอย่างจำนวนมากในห้องตัวอย่าง SBF-SEM และ FIB-SEM ได้ปริมาณที่เท่ากันในการศึกษานี้ และเวลาการได้มาของ SBF-SEM (2.5 ชั่วโมง) นั้นเร็วกว่าที่ได้รับจาก FIB-SEM (20 ชั่วโมง) ประมาณ 48 เท่า (ตารางที่ 1 ). อย่างไรก็ตาม FIB-SEM ได้จัดเตรียมภาพโครงสร้าง 3 มิติที่สร้างขึ้นใหม่ด้วยความละเอียด Z ที่สูงกว่า SBF-SEM ความแตกต่างของความละเอียดนี้เกิดจากความแตกต่างของความหนาของการตัด: 20 nm สำหรับ FIB-SEM และ 50 nm สำหรับ SBF-SEM
ผลลัพธ์เหล่านี้สรุปไว้ในตารางที่ 2 คุณภาพของภาพ SBF-SEM นั้นด้อยกว่าวิธีอื่นๆ อย่างไรก็ตาม SBF-SEM นั้นมีพื้นที่การสังเกตที่หลากหลายและความเร็วในการสังเกตที่ไม่มีใครเทียบได้ นอกจากนี้ ในตัวอย่างที่มีค่าการนำไฟฟ้าสูง คุณภาพของภาพจะดีขึ้นเนื่องจากแรงดันไฟเร่งจะเพิ่มขึ้นอีก สำหรับคุณสมบัติข้างต้น SBF-SEM เหมาะสำหรับตัวอย่างขนาดใหญ่มากที่มีค่าการนำไฟฟ้าสูงและสำหรับการวิเคราะห์ที่ครอบคลุม ตัวอย่างเช่น ตัวอย่างเนื้อเยื่อมีขนาดใหญ่ พื้นที่เรซินมีขนาดเล็ก และมีค่าการนำไฟฟ้าสูง จำเป็นต้องมีการวิเคราะห์ที่ครอบคลุมในการวิเคราะห์คอนเนกโทม
FIB-SEM ต้องการเวลาในการรับข้อมูลที่ยาวนานเมื่อเทียบกับ SBF-SEM อย่างไรก็ตาม FIB-SEM นั้นได้เปรียบที่ความละเอียด Z สูงมาก ข้อได้เปรียบนี้ใช้ได้เฉพาะกับพื้นที่สังเกตการณ์ที่แคบเท่านั้น FIB-SEM เหมาะสำหรับตัวอย่างขนาดเล็ก เช่น เนื้อเยื่อขนาดเล็กหรือทั้งเซลล์ FIB-SEM มีความสัมพันธ์กันสูงสำหรับการวิเคราะห์เชิงปริมาณ เนื่องจากปริมาณมากต้องการความละเอียดสูง นอกจากนี้ยังสามารถตัดกระดูกและโลหะได้ ทำให้สามารถวิเคราะห์เนื้อเยื่อแข็งและส่วนที่สัมผัสกันระหว่างโลหะกับเนื้อเยื่อได้ 3 มิติ
ในการตรวจเอกซเรย์แบบอาร์เรย์ ตัวอย่างยังคงอยู่หลังจากการสังเกต ในขณะเดียวกัน เทคนิคที่พัฒนาแล้วซึ่งสร้างส่วนต่อเนื่องและต้องใช้เวลาในการได้มาซึ่งนานขึ้นเพื่อเพิ่มความลึกที่สังเกตได้ Array tomography เหมาะสำหรับการวิเคราะห์รายละเอียดของปรากฏการณ์หายากในพื้นที่สังเกตขนาดใหญ่ เราคาดว่าซับสเตรตที่โปร่งใส เช่น แก้ว จะช่วยให้สามารถใช้วิธีการต่างๆ ที่สัมพันธ์กันได้ รวมถึงแสงสหสัมพันธ์และกล้องจุลทรรศน์อิเล็กตรอน (CLEM) Array tomography มีประสิทธิภาพในการสังเกตเซลล์ลอย แบคทีเรีย และเอ็มบริโอที่มีค่าการนำไฟฟ้าต่ำ ล้อมรอบด้วยเรซินรอบๆ ตัวอย่าง เนื่องจากตัวอย่างเป็นส่วนบางๆ บนซับสเตรตที่นำไฟฟ้า การชาร์จจะถูกระงับ และตัวอย่างสามารถสังเกตได้ด้วยแรงดันไฟฟ้าที่มีความเร่งสูง
รูปที่ 3 การเปรียบเทียบพื้นที่สังเกตและคุณภาพของภาพของแต่ละวิธี
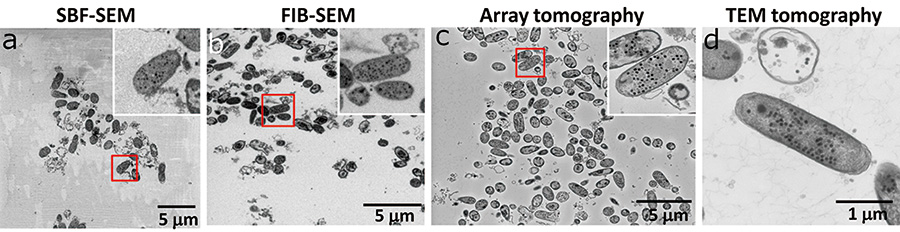
ใน SBF-SEM ชิ้นงานทดสอบจะถูกชาร์จอย่างง่ายดาย ทำให้คุณภาพของภาพแย่ลง เนื่องจากบล็อกเรซินที่มีการนำไฟฟ้าเพียงเล็กน้อยจะกินพื้นที่การสังเกตที่กว้าง (a) FIB-SEM ยังช่วยให้สังเกตพื้นที่เรซินบล็อกกว้างได้ แต่ไอออนของแกลเลียมที่พ่นบนพื้นผิวของตัวอย่างจะยับยั้งการชาร์จ (b) ในการตรวจเอกซเรย์แบบอาร์เรย์ เราสามารถสังเกตภาพที่มีอัตราส่วนสัญญาณต่อสัญญาณรบกวน (SN) สูงได้ เนื่องจากการชาร์จถูกระงับสำหรับส่วนที่เป็นชิ้นบางๆ ซึ่งติดตั้งบนแผ่นเวเฟอร์ซิลิคอนที่เป็นสื่อกระแสไฟฟ้าสูง (c) ในการตรวจเอกซเรย์ TEM เราสังเกตเพียงสิ่งเดียวเท่านั้น E.coli (ง)
รูปที่ 4 การเปรียบเทียบผลการสร้าง 3 มิติ

เราสร้างโครงสร้าง 3 มิติของ .ขึ้นใหม่ E.coli ติดเชื้อ T4 phages รูปภาพแสดงปริมาณของพื้นที่สังเกตการณ์ทั้งหมด (โฆษณา) ภาพการแบ่งกลุ่มของหนึ่ง E.coli เซลล์ที่ติดเชื้อ T4 phages (สีเขียว), T4 phages ที่เติบโตใน E.coli (สีส้ม) และผู้ติดเชื้อ (สีชมพู) (เอ๊ะ) ภาพการแบ่งกลุ่มของ T4 phages หนึ่งตัวใน E.coli (อิล)
ตารางที่ 2 สรุปคุณสมบัติของการวิเคราะห์โครงสร้าง 3 มิติ ด้วยกล้องจุลทรรศน์อิเล็กตรอน
| SBF-SEM | FIB-SEM | เอกซเรย์อาร์เรย์ | TEM เอกซเรย์ | |
|---|---|---|---|---|
| ขอบเขตการมองเห็น (X,Y) | มากกว่า 100 μm | 1~100 μm | 100 ไมโครเมตร | น้อยกว่า 5 ไมโครเมตร |
| ระยะชัดลึก (Z) | ลึกมาก | ลึก | แคบ | แคบมาก |
| ความละเอียด 3 มิติ (Z) | นาโนเมตร 50 | นาโนเมตร 20 | นาโนเมตร 70 | นาโนเมตร 1 |
| เวลาที่ได้มา | บางทศวรรษของเรา | บางวัน | บางวัน | 1~2 ชั่วโมง |
| ความพยายามที่จะได้มา | น้อย | น้อย | มหาศาล | น้อย |
| ตัวอย่างที่เหมาะสมที่สุด | เนื้อเยื่อสมอง | เนื้อเยื่อ เซลล์เพาะเลี้ยง | เนื้อเยื่อ ตัวอ่อน เซลล์เพาะเลี้ยง | ออร์แกเนลล์ ไวรัส |
| แอปพลิเคชั่นที่เหมาะสมที่สุด | Conectome การวิเคราะห์อย่างละเอียดถี่ถ้วน | การสังเกตทางสัณฐานวิทยา การวิเคราะห์เชิงปริมาณ | การวิเคราะห์เชิงปริมาณ CLEM | การสังเกตทางสัณฐานวิทยา |
การวิเคราะห์ภาพการสร้าง 3 มิติ
① ความสัมพันธ์ของสัณฐานวิทยาของ E.coli ที่ติดเชื้อโดย T4 phages และ internal phage particle
SBF-SEM ก็เพียงพอที่จะวิเคราะห์สัณฐานวิทยาของ E.coli และการกระจายของ T4 phage เราวิเคราะห์ความสัมพันธ์ของสัณฐานวิทยาของ E.coli และฟาจภายในโดยใช้ผลลัพธ์ของ SBF-SEM ก่อนอื่นเราตรวจสอบความสัมพันธ์ระหว่างปริมาณของ E.coli และจำนวนเฟส (รูปที่ 5 ก) ผลลัพธ์แสดงให้เห็นว่าจำนวนสูงสุดของฟาจภายในใน E.coli มีประมาณ 280 อนุภาคและไม่มี E.coli ที่มีอนุภาคฟาจมากขึ้นสามารถสังเกตได้ เราพิจารณาว่าเมื่อจำนวนอนุภาคฟาจเพิ่มมากขึ้น การเพิ่มขึ้นนี้ทำให้เกิด E.coli ที่จะระเบิด จำนวนอนุภาคฟาจที่มีอยู่มีแนวโน้มเพิ่มขึ้น เมื่อปริมาตรของ E.coli เพิ่มขึ้น อย่างไรก็ตาม ความสัมพันธ์นี้ไม่เป็นเชิงเส้น มีจำนวนอนุภาคฟาจแตกต่างกันแม้ในปริมาตรเดียวกัน E.coli (B และ C, D, E ในรูปที่ 5 a) เราจึงเลือกคุณลักษณะ E.coli (AE ในรูปที่ 5 a, c) เลือกแล้ว E.coli เซลล์มีดังนี้ พวกมันมีปริมาตรน้อยและมีอนุภาคฟาจน้อย (A, B ในรูปที่ 5 a, c) อันต่อไปก็มีปริมาตรน้อย แต่ก็มีอนุภาคฟาจจำนวนมาก (CE ในรูปที่ 5 a, c) อีกอันหนึ่งมีปริมาตรมากและมีอนุภาคฟาจจำนวนมาก (F ในรูปที่ 5 a, c) E.coli เซลล์ถูกแบ่งออกเป็นสามส่วนตามทิศทางแกนยาว ส่วนตรงกลาง ส่วนตรงกลาง และส่วนด้านข้าง เราวิเคราะห์ว่าส่วนใดของอนุภาคฟาจ T4 ถูกแปลเป็นภาษาท้องถิ่น (รูปที่ 5 b) อนุภาคฟาจส่วนใหญ่ถูกจำกัดตำแหน่งไว้ที่ส่วนตรงกลาง เมื่ออนุภาคฟาจมีน้อย อนุภาคฟาจที่แปลเป็นภาษาท้องถิ่นในส่วนตรงกลางถูกกระจายจากส่วนตรงกลางไปยังส่วนข้าง เมื่ออนุภาคฟาจภายในเพิ่มขึ้น นอกจากนี้ เนื่องจากปริมาณของ E.coli เพิ่มขึ้น อนุภาคฟาจในส่วนตรงกลางลดลงมากขึ้น และอนุภาคฟาจที่แปลเป็นภาษาท้องถิ่นในส่วนด้านข้างเพิ่มขึ้น ต่อไปเราเน้นที่รูปร่างของ E.coli . รูปร่างของ E.coli ที่มีอนุภาคฟาจ T4 น้อยมีรูปร่างเหมือนแท่ง (รูปที่ 5 c B) เมื่ออนุภาคฟาจภายใน T4 เพิ่มขึ้น E.coli ขยายไปยังทิศทางแกนสั้นและรูปร่างก็กลายเป็นรูปร่างคล้ายลำกล้อง (รูปที่ 5 c CE)
รูปที่ 5 ความสัมพันธ์ของสัณฐานวิทยาของ E.coli และฟาจที่เกี่ยวข้อง
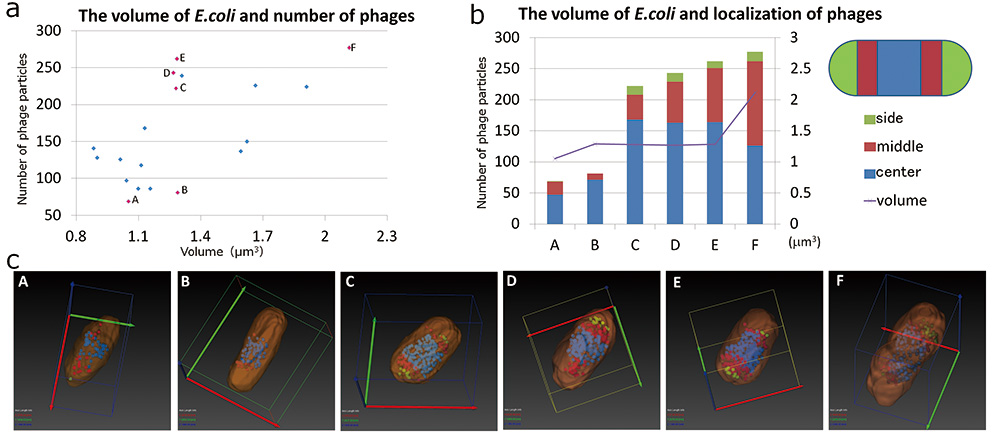
กราฟด้านซ้ายแสดงความสัมพันธ์ของปริมาตรของ E.coli ติดเชื้อ T4 phage และจำนวน T4 phages ที่เกี่ยวข้อง (a) เลือกลักษณะเด่น XNUMX ประการ E.coli เซลล์ (จุดสีชมพูในกราฟด้านซ้าย AF) สัณฐานวิทยาของ6 .เหล่านี้ E.coli เซลล์ที่เกี่ยวข้องกับฟาจ T4 ถูกมองเห็นโดยการแบ่งส่วน (c) กราฟด้านขวาแสดงความสัมพันธ์ของปริมาตรของ E.coli และการกระจายของ T4 phage (b)
② ความสัมพันธ์ระหว่างชนิดของฟาจและการกระจายของฟาจใน E.coli
T4 phage สามารถรับรู้ได้โดยการวิเคราะห์โครงสร้าง 3 มิติด้วย SEM เนื่องจากมีคอนทราสต์สูงใน E.coli . อย่างไรก็ตาม λ phage และφ174 phage นั้นยากต่อการจดจำใน E.coli เพราะมีคอนทราสต์ต่ำใน E.coli , และฟาจฟาจมีขนาดเล็กเกินไป (รูปที่ 174 e, c, f) ดังนั้น การเปรียบเทียบของ E.coli ติดเชื้อโดย T4 phage, λphage และφ174 phage โดยใช้ภาพการสร้าง 3 มิติโดย Array tomography ก่อนอื่นเราเน้นที่สัณฐานวิทยาของ E.coli . รูปร่างของ E.coli การติดเชื้อจากλphageเปลี่ยนจากโครงสร้างคล้ายแท่งเป็นโครงสร้างคล้ายลูกบอล เราแยกจากกัน E.coli ฟาจที่ติดเชื้อเป็นสามชั้น ชั้นใน ชั้นกลาง และชั้นนอก และตรวจสอบว่าฟาจใดถูกแปลเป็นภาษาท้องถิ่นในชั้นใด (รูปที่ 6 ก) ผลการศึกษาพบว่า T4 phage และφ174 phage กระจายตัวใน E.coli โดยปราศจากอคติ อย่างไรก็ตาม อนุภาคฟาจเกือบทั้งหมดถูกแปลเป็นภาษาท้องถิ่นที่ชั้นนอก T4 phage และφ174 phage เป็น lytic phage ทันทีที่ติดเชื้อ E.coli พวกมันเริ่มแพร่ขยายในเซลล์เจ้าบ้านและเซลล์เจ้าบ้านแตกออก (lyse) ในทางกลับกัน λphage เป็น phage ที่มีอุณหภูมิปานกลาง เมื่อติดเชื้อ E.coli และฉีดจีโนมเข้าไปในเซลล์เจ้าบ้าน พวกมันจะไม่สลายทันที หลังจากการติดเชื้อ พวกมันจะแทรกจีโนมของพวกมันเข้าไปในจีโนมของโฮสต์และทำตัวเป็นส่วนหนึ่งของโฮสต์ (lysogenization) อย่างไรก็ตาม ตัวยับยั้งซึ่งยับยั้งการเพิ่มจำนวนของฟาจถูกปิดใช้งานโดยการเปลี่ยนแปลงของสภาพแวดล้อม ฟาจเริ่มที่จะเพิ่มจำนวนและโฮสต์ไลซ์ ความแตกต่างในการเปลี่ยนแปลงทางสัณฐานวิทยาที่สังเกตได้และการแปลความหมายของฟาจอาจมาจากพื้นที่การงอกขยาย ไลติกฟาจเพิ่มจำนวนขึ้นในพื้นที่ที่ติดเชื้อทันที และฟาจที่มีอุณหภูมิพอเหมาะจะแพร่ขยายบนจีโนมเจ้าบ้านฟาจไม่แสดงการกระจายแบบเอนเอียงในสามชั้น แต่มีการแปลเป็นภาษาท้องถิ่นใน ส่วนเฉพาะ (รูปที่ 174 ง) ในการทดลองนี้ เราไม่สามารถระบุสถานที่ที่ติดเชื้อแห่งแรกได้ แต่สถานที่ที่แปลอาจเกี่ยวข้องกับสถานที่แรกที่ติดเชื้อ
รูปที่ 6 การกระจายเฟส 3 มิติใน E.coli
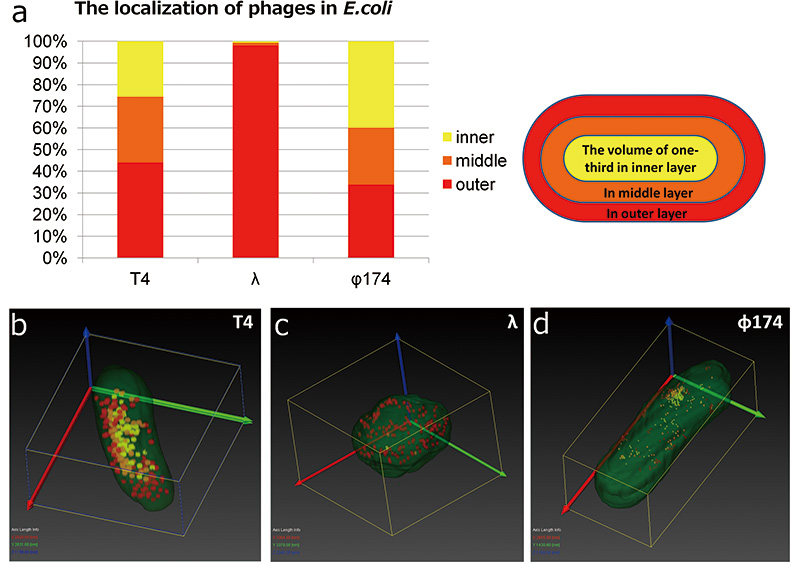
เราเปรียบเทียบการกระจายของฟาจจากผลลัพธ์ของการสร้างโครงสร้าง 3 มิติใหม่ของ T4 phage, λphage และ φ174 phage ในรูปแบบ Array tomography เราแยกจากกัน E.coli เป็นสามส่วน; บริเวณด้านใน ตรงกลาง และด้านนอก แล้วนับจำนวนฟาจที่เกี่ยวข้องในแต่ละส่วน T4 phages และ φ174 phages มีอคติเพียงเล็กน้อยในการกระจาย อย่างไรก็ตาม เกือบจะλphages แปลเป็นภาษาท้องถิ่นที่พื้นที่ด้านนอก (a) E.coli ที่ติดเชื้อλฟาจเปลี่ยนรูปร่างจากก้านเป็นทรงกลม (c)
สรุป
วิธีการสร้าง 3D ใหม่แต่ละวิธีด้วย SEM มีจุดแข็งและจุดอ่อนของตัวเอง ทั้งนี้ขึ้นอยู่กับการเตรียมตัวอย่างและสภาวะการได้มา เช่น แรงดันไฟฟ้าแบบเร่ง ใน SBF-SEM และ FIB-SEM การเก็บข้อมูลจะดำเนินการโดยอัตโนมัติ ในทางกลับกัน หลังจากการวิเคราะห์การแบ่งส่วนมักจะดำเนินการด้วยตนเอง การแบ่งส่วนซึ่งแยกเนื้อเยื่อและบริเวณที่สนใจมักดำเนินการตามคอนทราสต์ของภาพ อย่างไรก็ตาม ตัวอย่างทางชีววิทยามีความแตกต่างกันเล็กน้อย และบางครั้งความแตกต่างเพียงอย่างเดียวอาจไม่สามารถกำหนดการแบ่งส่วนได้ เป็นไปได้ที่จะแบ่งส่วนด้วยสัณฐานวิทยาเป็นจุดสังเกตโดยการรักษาความละเอียดสูงของภาพ และด้วยเหตุนี้จึงช่วยปรับปรุงประสิทธิภาพการแบ่งส่วน SBF-SEM และ FIB-SEM จำเป็นต้องแนะนำเครื่องมือเฉพาะ ในขณะที่การตรวจเอกซเรย์แบบอาร์เรย์สามารถเริ่มต้นด้วย SEM แบบเดิมและ ultramicrotome แต่จำเป็นต้องมีทักษะบางอย่าง การเลือกวิธีที่จะใช้เป็นกลยุทธ์สำคัญในการส่งเสริมการวิจัยอย่างมีประสิทธิภาพ เราหวังว่าบทความนี้จะเป็นประโยชน์สำหรับนักวิจัยที่กำลังวางแผนที่จะเริ่มการวิเคราะห์ 3 มิติด้วยกล้องจุลทรรศน์อิเล็กตรอน
กิตติกรรมประกาศ
ผู้เขียนขอขอบคุณชาว JEOL ที่สนับสนุนการศึกษาครั้งนี้ H. Nishioka สำหรับการสนับสนุนผู้บริหารในการศึกษานี้ M. Suga สำหรับการสนับสนุนการเก็บข้อมูลของ Array tomography, H. Matsushima สำหรับการสนับสนุนการเก็บข้อมูลด้วย FIB-SEM, Y. Yamaguchi สำหรับการอภิปรายที่เป็นประโยชน์และคำแนะนำทางเทคนิคของ SBF- SEM, K. Hasumi สำหรับการสนับสนุนการแบ่งส่วน ผู้เขียนขอขอบคุณสถาบันเทคโนโลยีและการประเมินแห่งชาติ สถาบันวิทยาศาสตร์และเทคโนโลยีอุตสาหกรรมขั้นสูงแห่งชาติ E.coli และฟาจ
อ้างอิง
- Knott G, Marchman H, Wall D, Lich B. กล้องจุลทรรศน์อิเล็กตรอนแบบส่องกราดส่วนอนุกรมของเนื้อเยื่อสมองผู้ใหญ่โดยใช้การกัดลำแสงไอออนแบบโฟกัส Neurosci J. (2008)
- มิเชวา เคดี, สมิธ เอสเจ. Array tomography: เครื่องมือใหม่สำหรับการถ่ายภาพสถาปัตยกรรมโมเลกุลและโครงสร้างพื้นฐานของวงจรประสาท เซลล์ประสาท. (2007)
- Deerinck, TJ & Bushong, Eric & Thor, A & Ellisman, มาร์ค วิธีการ NCMIR สำหรับ 3D EM: โปรโตคอลใหม่สำหรับการเตรียมตัวอย่างทางชีวภาพสำหรับกล้องจุลทรรศน์อิเล็กตรอนแบบส่องกราดใบหน้าแบบอนุกรม Nat Center Microsc ภาพความละเอียด. 6-8. (2010).
- Denk W, Horstmann H. กล้องจุลทรรศน์อิเล็กตรอนแบบส่องกราดแบบบล็อกใบหน้าแบบอนุกรมเพื่อสร้างโครงสร้างนาโนเนื้อเยื่อสามมิติขึ้นใหม่ PLOS Biol. (2004)
- Schindelin J, Arganda-Carreras I, Frise E, Kaynig V, Longair M, Pietzsch T, Preibisch S, Rueden C, Saalfeld S, Schmid B, Tinevez JY, ดีเจสีขาว, Hartenstein V, Eliceiri K, Tomancak P, Cardona A. ฟิจิ: แพลตฟอร์มโอเพ่นซอร์สสำหรับการวิเคราะห์ภาพทางชีววิทยา วิธีการของแนท. (2012)
สินค้าที่เกี่ยวข้อง
กล้องจุลทรรศน์อิเล็กตรอนแบบส่องผ่าน (TEM)
การแก้ปัญหาตามสาขา
คุณเป็นผู้เชี่ยวชาญทางการแพทย์หรือบุคลากรที่เกี่ยวข้องกับการรักษาพยาบาลหรือไม่?
ไม่
โปรดทราบว่าหน้าเหล่านี้ไม่ได้มีวัตถุประสงค์เพื่อให้ข้อมูลเกี่ยวกับผลิตภัณฑ์แก่ประชาชนทั่วไป
